지난 포스트에서 예고한대로 오늘은 NEAT (Neuroevolution of Augmenting Topologies)에 대해 알아보겠습니다. Neuroevolution (NE)이라는 방법론적인 차이는 있지만 network topology learning은 결국 AutoML과 NAS (Neural Architecture Search)까지 이어지는 개념이기 때문에 흥미로운 주제가 될 것 같습니다. 게다가 개인적으로 NE의 열렬한 팬이기도 합니다.
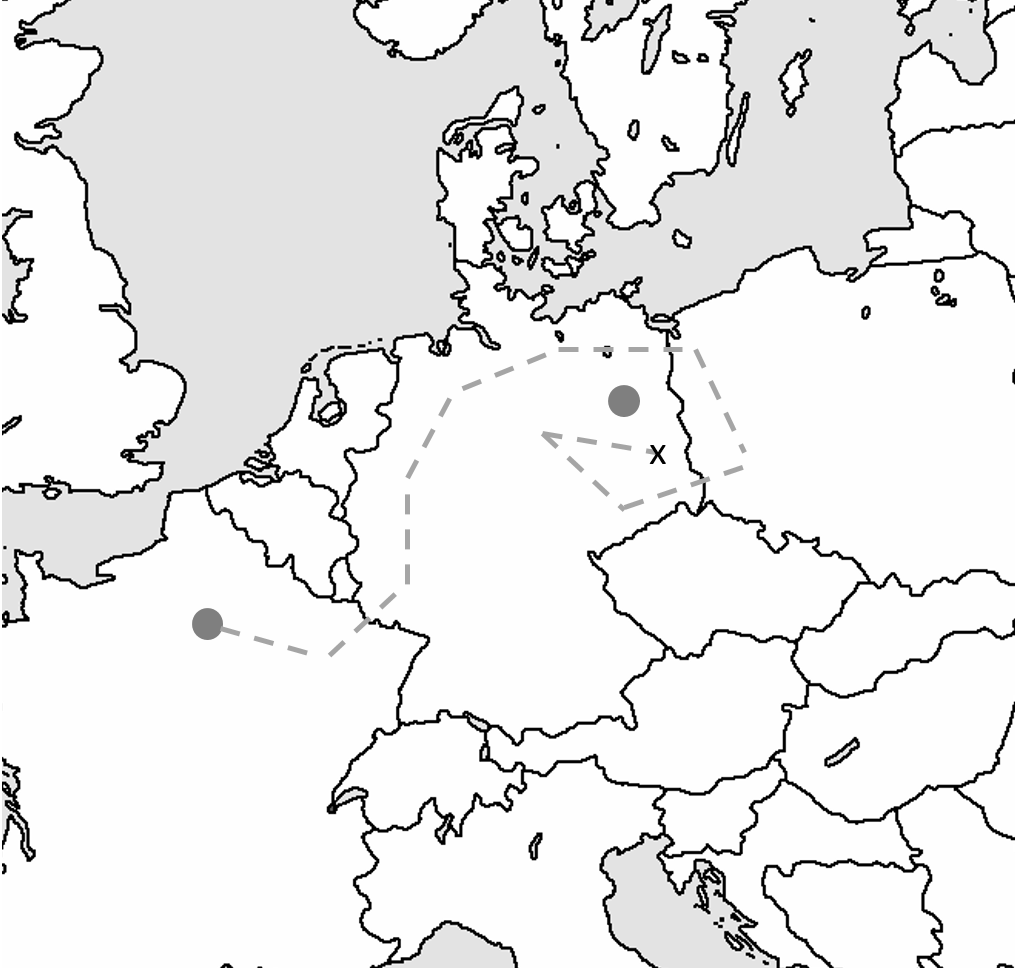
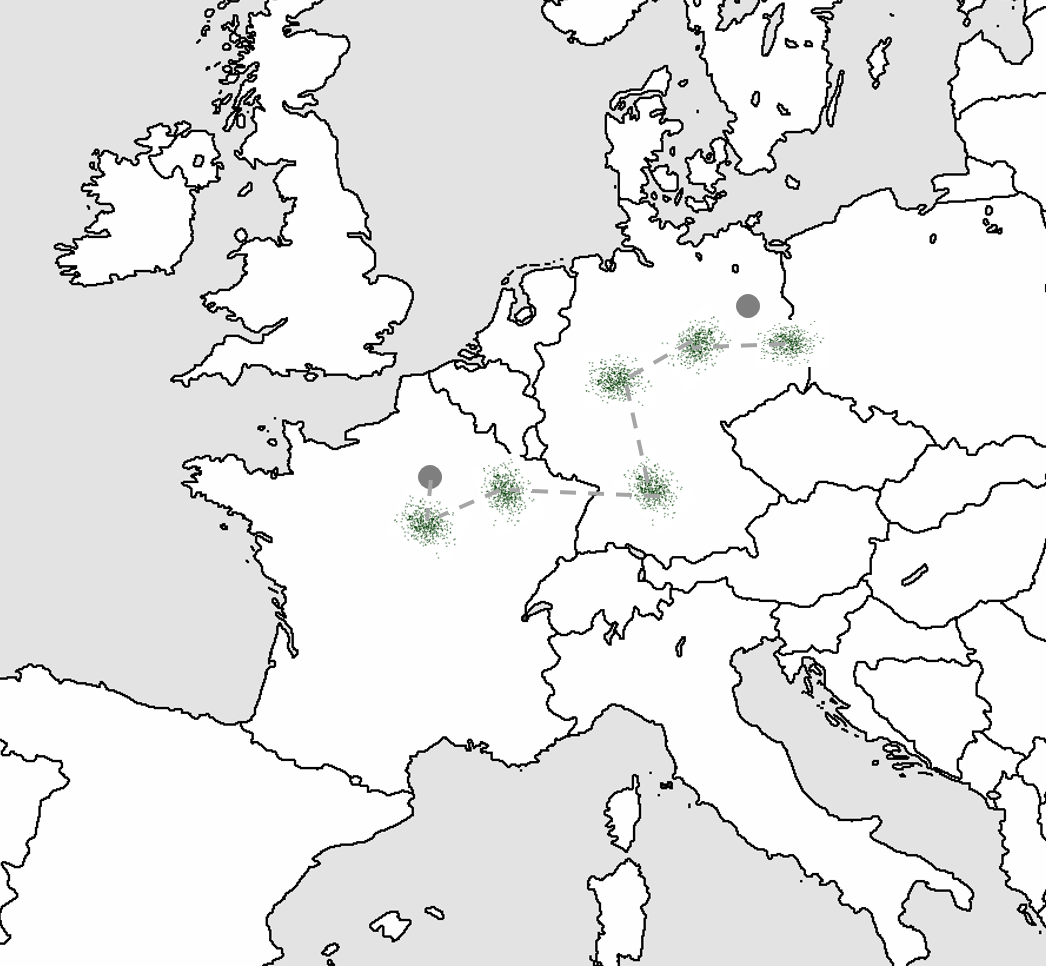
예전에 다른 포스트에서 한 번 언급했듯이 다른 innovation과 마찬가지로 NE도 90년대 말 minor 혁신들을 통해서 지속적인 성능 개선이 이루어져왔습니다 (예를 들면 ESP 같은). NE가 reinforcement learning (RL) task에 좋은 성능을 보인다는건 지난번 포스트에서도 말씀을 드렸습니다. 사실 다른 종류의 학습, 예를 들면 supervised learning에도 NE를 적용하지 못할 이유는 없지만, problem space 또는 optimisation surface가 연속적이고 미분가능하게 ‘well define’된 경우라면 굳이 population을 만들어서 주변을 탐색하는 일은 낭비가 될 가능성이 크겠죠. 이런 경우라면 gradient descent를 이용한 방식이 훨씬 효과적일 것입니다. 아래 그림은 Paris에서 출발하여 Berlin에 도착하기 라는 가상의 task를 만들어서 gradient descent 방법과 NE의 탐색 방법이 어떻게 다른지 재미있게 그림으로 그린 예시입니다 (이미지 출처). Gradient descent가 매 iteration마다 일정한 거리를 가면서 Berlin이 어디인지를 물어 방향을 고쳐잡는 방식이라고 한다면, NE는 매 generation마다 주변의 여러 사람들에게 Berlin 방향을 물어 탐색하는 방식이라 할 수 있습니다. 아주 정확한 설명은 아니지만 감을 잡으시는데는 도움이 될 것 같습니다. Gradient descent와 neuroevolution의 solution 탐색 장단점에 대해서는 이후 Uber AI의 neuroevolution research에 대해 이야기할 때 다시 한 번 다루도록 하겠습니다.
| Gradient Descent | Neuroevolution |
|---|---|
 |
 |
Search space의 차원이 높은 경우 경험을 바탕으로 가치함수를 업데이트하는 방식으로 학습하는 RL보다 behavior를 직접 학습하는 NE가 성능이 더 좋을 것입니다. 음, 더 잘 이해하려면 RL에 대한 기본 지식이 있으면 좋은데, 일단은 강화학습에 대해 잘 정리된 블로그를 링크하고 넘어가도록 하고, 기회가 되면 다시 한 번 돌아보도록 하겠습니다 (링크, 열심히 공부하시는 분들이 많습니다 ^^). 이러한 NE의 가능성에도 불구하고 기존의 방식은 네트워크의 topology는 고정한 채 weight만 학습하는 경우가 대부분이었습니다. 2001년에 NEAT가 발표되기 전 네트워크의 구조를 학습하려는 시도가 전혀 없진 않았지만 썩 좋은 결과를 내지는 못하고 있었습니다. ‘Topology(structure)를 weight와 동시에 학습하는 것이 weight만 학습하는 것과 비교해 성능이 더 좋은가?’ 라는 질문에 답하기 위해서는 해결해야 할 문제들이 몇 가지 있습니다:
- Topology를 효과적으로 나타내고, crossover 및 mutation도 적용시킬 수 있는 genotype이 있는가?
- Topological evolution이 최적화될 수 있도록 보호해 줄 방법이 있는가?
- Topological evolution으로 인해 네트워크가 과다하게 복잡해지는 것을 효과적으로 조절할 방법이 있는가?
NEAT는 이 세가지 질문에 대한 해답을 (그것도 biological grounding을 가진 해답을) 제시하면서 뜨거운 반응을 불러일으켰고, 이후 업그레이드 된 버전(HyperNEAT)까지 다양한 언어로 개발되어 여러 RL application에 적용되었습니다 (Stanley 교수님의 NEAT 웹사이트로 NEAT의 확장에 대한 더 자세한 설명은 생략합니다). 이제 위의 세 질문에 대한 NEAT의 답변을 살펴보도록 하시죠.
Topology를 효과적으로 나타내고, crossover 및 mutation도 적용시킬 수 있는 genotype이 있는가?
Competing Conventions Problem
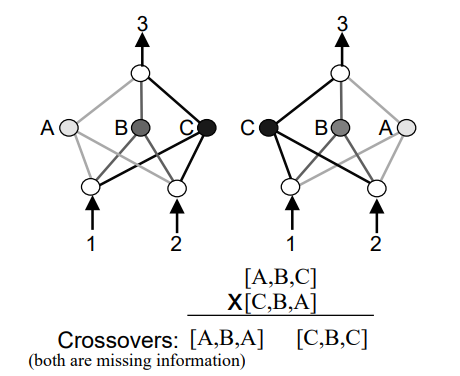
지난 포스트에서 보셨듯이 gene의 조합인 chromosome으로 이루어진 기존 NE의 genotype으로는 topology를 표현하는 것이 불가능했지요. NEAT에서는 genotype을 네트워크의 node를 나타내는 node gene의 list와 네트워크의 weight를 나타내는 connection gene의 list로 구성하는 direct encoding 방식을 택하였습니다 (살짝 off the topic이지만, indirect encoding은 compact한 representation이 가능하다는 장점이 있지만 구현과 해석이 복잡하다는 단점이 있습니다. NEAT에 indirect encoding 방법을 적용한 HyperNEAT가 나중에 개발되어서 NEAT보다 훨씬 큰 ANN을 다룰 수 있게 됩니다). Node gene과 connection gene의 list로 phenotype(network)을 바로 만들어낼 수는 있게 되었지만 이런 structure 정보를 가진 genotype으로 crossover를 하려니 문제가 있습니다. Competing Conventions Problem 이라 부르는 문제인데요, 일반적인 non-topological NE에도 존재하던 문제였습니다. 아래의 예를 보시면 Competing Conventions Problem 에 대한 직관적인 이해가 가능할 것 같습니다. Hidden layer의 node들의 순서만 다른, 사실상 거의 동일한 기능을 수행하는 네트워크 2개가 parents로 선택되어 crossover를 수행하게 되면 자칫 학습된 task 수행능력을 잃은 ‘멍청한’ offspring을 만들어내게 될 경우가 생기는거죠. Hidden layer A, B, C가 task 수행에 필요하여 학습된 상태인데, [A, B, C]와 [C, B, A] 네트워크를 mating 시켜 [A, B, A]와 [C, B, C]가 만들어지면 next generation의 fitness가 곤두박질 칠 것입니다.
Innovation Number (Historical Marking)
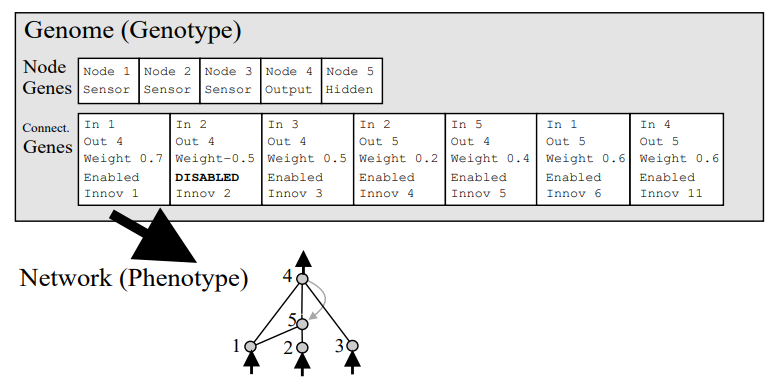
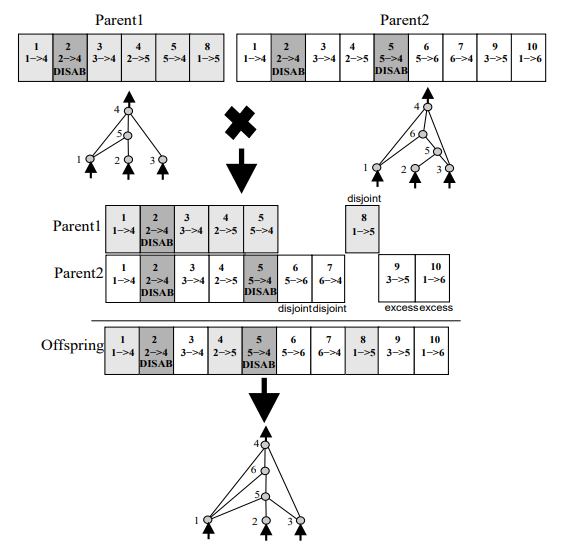
이러한 Competing Conventions Problem 은 네트워크의 특정 sub-structure가 task 수행에 있어 특정 기능(sub-function)을 담당하는 topological NE에서는 이 문제가 더 심각해질 것입니다. NEAT에서는 NE답게 biologically-inspired 솔루션을 제공하는데요, 염색체가 crossover될 때 RecA라는 단백질에 의해 ‘homologous한’ gene들이 정렬된다는 점에 착안하여 NEAT chromosome의 topology를 담당하는 connection gene들에 매 structural update가 일어날 때마다 increment되는 innovation number 를 부여해서 각 node와 connection의 history를 marking하면서 homologous gene을 구분할 수 있게 한 것입니다. 무슨 말인지 이해가 잘 안되면 그림을 보면 되지요. 우선 아래의 그림을 보면 NEAT의 sample chromosome을 통해 genotype과 phenotype이 어떻게 mapping되는지를 알 수 있습니다. 5개의 node gene과 6개의 connection gene(1개는 DISABLED)은 네트워크가 5개의 노드와 6개의 edge로 구성되어있다는 것과, 각 edge가 어느 node들을 어느 방향으로 (예를 들어 맨 마지막 connection gene은 node 4에서 node 5 방향으로 연결되는 recurrent connection을 뜻하겠지요?) 연결되는지를 보여줍니다. 각 connection gene의 weight는 조금 후에 설명하겠습니다. Node gene에는 해당 node가 sensor (input), hidden, output 중 어떤 layer에 속하는지 다소 redundant한 정보도 담고 있네요. Connection gene의 맨 아래에 innovation number가 할당되어 있는 것을 볼 수 있는데요, uniformly increasing한 값이기 때문에 모든 connection gene은 unique한 innovation number를 가지고 있습니다. EN/DISABLE bit은 해당 edge가 활성화되었는지를 나타내는데요, 뒤의 내용을 좀 더 읽다보면 알게 되겠지만, 2-to-4 연결이었던 Innov 2 connection gene이 DISABLE 되어있고 2-to-5, 5-to-4 연결을 맡은 Innov 4, Innov 5 connection gene들이 Innov 2 connection gene보다 더 큰 innovation number를 가진 것으로 보아, 원래 2-to-4 direct connection이 존재하였다가 어느 시점에 crossover나 mutation에 의해 5번 node가 2와 4 사이에 생겨나게 되었고, 1-to-5 connection gene이 Innov 6 값을 가지고 있는 것으로 보아, 5번 node가 2와 4 사이에 추가된 뒤에 1-to-5 connection이 새로 추가되었음을 알 수 있습니다. 헷갈려도 참고 좀 더 보시죠.
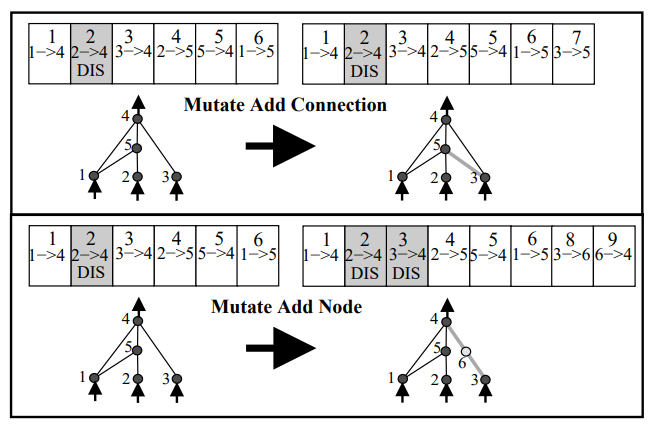
Topological Mutation
아래 그림은 NEAT의 topological mutation을 나타냅니다. Crossover보다 간단해서 먼저 설명합니다. 물론 mutation 중 topology 변화 없이 weight 값만 바뀌는 경우도 있겠지만 이건 trivial하니 설명이 필요한 topological mutation만 그림으로 보였습니다. 그림이 직관적이라 별다른 설명이 필요없긴 하지만, 보시면 topological mutation은 connection이 추가되는 경우와 node가 추가되는 경우로 구분할 수 있습니다. 위쪽이 mutate add connection인데요, 굳이 표시하지는 않았지만 chromosome의 node gene은 변화가 없으니 그대로일테고 3-to-5 connection만 connection gene list에 추가되고 +1된 innovation number가 할당되었습니다. 아래쪽은 mutation add node인데요, 역시 그림에는 없지만 이 경우에는 node gene list에 6번 node가 추가됩니다. 3-to-4 connection 사이에 node 6이 추가된 것이기 때문에 3-to-6, 6-to-4 connection이 connection gene list에 새로운 innovation number를 가지고 추가되고, 기존의 3-to-4 direct connection은 DISABLE된 것을 알 수 있습니다. 이 그림에서는 connection gene의 weight도 생략되어있는데요, add connection의 경우는 새 connection에 random 값이 할당되고, add node로 인해 기존 connection이 분기되는 경우는 1과 기존 connection(DISABLE되는 connection)의 weight를 새로 분기된 2개의 connection의 weight로 할당해서 structural modification으로 인한 fitness의 ‘충격’을 최소화시켰습니다. 이 그림의 예를 들어 설명하자면 Innov 8의 weight로 1이 할당되고, Innov 9의 weight에는 Innov 3의 weight를 할당하게 되는거죠. Connection gene의 DISABLE 여부도 probabilistic하게 변경할 수 있습니다. 걱정했던 것보다는 간단하죠?
만약 mutation을 통해 동일한 형태의 structural update가 한 번 이상 발생하는 경우, 뒤에 만들어지는 structure가 기존 generation pool에 있는지 확인하고, 매칭되는 structure가 없는 경우에만 innovation number를 추가적으로 increment시키고 node나 connection gene list를 업데이트 해줍니다. 매칭되는 structure가 있다는 말은 지금과 동일한 structure가 이번 mutation에서 조금 전에 만들어졌다는 뜻이므로 (동일한 history를 가진다는 의미이므로) innovation number를 추가로 변경할 필요 없이 node나 connection gene list를 업데이트하면 될 것이고, 결국 weight는 다를 수 있지만 innovation number를 포함한 다른 값들은 완전히 동일한 chromosome이 만들어지게 되는 것입니다.
반대로 connection이나 node가 줄어드는 mutation은 없는지 혹시 궁금하실 수 있는데요, 이런 경우는 없습니다. NEAT는 task를 수행할 수 있는 minimal한 topology를 학습하는 것을 목표로 하기 때문에 최소한의 structure로 초기화를 한 후 topology를 늘려가는 방식으로 학습을 하기 때문입니다.
Topological Crossover
이제 대망의 crossover입니다. 이 그림만 이해하면 NEAT의 주요 설명은 끝난다고 볼 수 있습니다! 여차저차해서 connection gene list가 아래와 같이 생긴 두 parent가 elitism으로 선택되어 crossover를 통해 offspring을 만들게 되었습니다. 가운데 부분에 innovation number를 이용하여 앞서 설명드렸던 homologous한 gene의 정렬(synapsis라고 합니다)이 이루어진 것을 보실 수 있는데요, 두 네트워크가 상당히 다르게 생겼지만 innovation number를 통해 gene을 어떻게 align시켜야 하는지 쉽게 파악할 수 있습니다. 정렬된 상태에서 multi-point crossover가 수행된 것으로 보이는데요, line-up된 일부 connection gene 아래 ‘disjoint’와 ‘excess’라고 표기가 되어있습니다. NEAT의 crossover에서는 matching되는 gene(Innov 1$\sim$5)끼리는 multi-point crossover(random 선택)를, disjoint와 excess gene의 경우는 두 parent 중 fitness가 더 높은 쪽을 따르도록 (fitness가 같다면 얘들도 random) 정하였습니다. 그림에서 Offspring이 Innov 6, 7, 9, 10번 connection gene을 가지고 있는 것으로 보아 ${Parent1}_{fitness}$ ≤ ${Parent2}_{fitness}$ 란걸 알 수 있겠네요. Offspring의 node gene list는 crossover된 connection gene list에 따라 구성하면 됩니다.
Topological evolution이 최적화될 수 있도록 보호해 줄 방법이 있는가?
Crossover나 mutation으로 생겨난 topology가 변한 offspring이 처음부터 앞 generation에서 fitness가 높아 선택된 parent보다 더 성능이 좋을 가능성은 별로 없습니다. 이러한 새로운 sub-structure(niche 라고도 합니다)가 task performance에 도움이 될 지는 추가적인 학습을 통해 해당 부분의 weight를 전체 weight와 같이 학습하고 최적화를 시켜봐야 알겠지요. 하지만 기존 NE 알고리즘으로는 niche 가 생기자마자 다른 개체들과 무제한 경쟁에 들어가게 되어, 생기자마자 도태되는 현상이 반복되는걸 피할 방법이 없었습니다. 자연에서의 speciation(종분화)은 종 안에서 돌연변이 등으로 발생된 새로운 niche 를 가진 개체군이 다른 개체군과 selective pressure를 받으면서 경쟁하다가 genotypic 또는 phenotypic 분기를 겪게 됩니다. 이 점에 착안해 NEAT에서는 chromosome의 similarity measure를 통해 compatible한 종을 구분하고, 이 안에서만 niche 가 경쟁할 수 있도록 함으로써 새로이 발견된 structure가 성능 좋은 다른 종(dissimilar한 네트워크)으로부터 생존의 위협을 받지 않게끔 하였습니다. 여기서도 Innovation Number가 활용됩니다. Similarity는 앞서 보셨던 disjoint gene의 숫자($D$), excess gene의 숫자($E$), 그리고 matching gene의 weight값의 차이($\overline{W}$)로 계산합니다:
$\delta = \frac{c_1E}{N} + \frac{c_2D}{N} + c_3\cdot\overline{W}$
이 similarity measure로 population 내 species의 구분이 가능해졌는데요, 이를 이용해 population 내 species의 다양성을 확보해주기 위해 각 individual의 fitness를 다음과 같이 조정해줍니다:
$f_i’ = \frac{f_i}{\sum_{j=1}^{n}sh(\delta(i, j))}$
$sh(\cdot) = 0$ if $\delta(i, j) < \delta_{threshold}$ and $sh(\cdot) = 1$ otherwise
Topological evolution으로 인해 네트워크가 과다하게 복잡해지는 것을 효과적으로 조절할 방법이 있는가?
NEAT는 uniform minimal structure를 가지는 population에서부터 진화를 시작하여 problem solving에 필요한 만큼만의 구조적 변화를 일으키는 방식으로 이 문제를 간단히 해결합니다. 앞서 mutation에서 historical marker를 사용해 동일한 structure가 다른 innovation number를 가지고 중복적으로 생기는 걸 막았던 것도 minimal topology 증가를 도와주는 부분이라고 할 수 있겠죠.
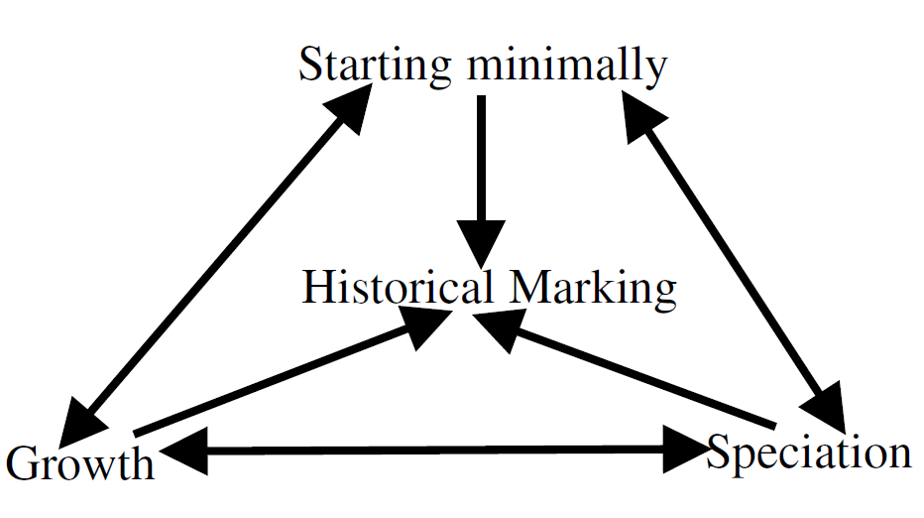
이상 살펴본 NEAT의 핵심은 Innovation number를 이용한 historical marking입니다. 이를 통해 효과적인 chromosome expression이 가능했고, Competing Conventions Problem , mutation과 crossover의 문제, 그리고 speciation과 minimal topology의 모든 문제들을 해결할 수 있었습니다.
이후 NEAT는 hypercube 기반의 indirect encoding을 적용한 HyperNEAT 등으로 연구가 확장되었고 다양한 강화학습 문제에 경쟁력있는 솔루션임을 보였습니다. 예전에 읽었던 논문을 리뷰하면서 개인적으로는 즐거운 시간을 보냈네요. Demo 영상 몇 개 보시고 마무리하겠습니다. 다음 포스트부터는 Uber AI의 Neuroevolution research로 넘어가보죠.